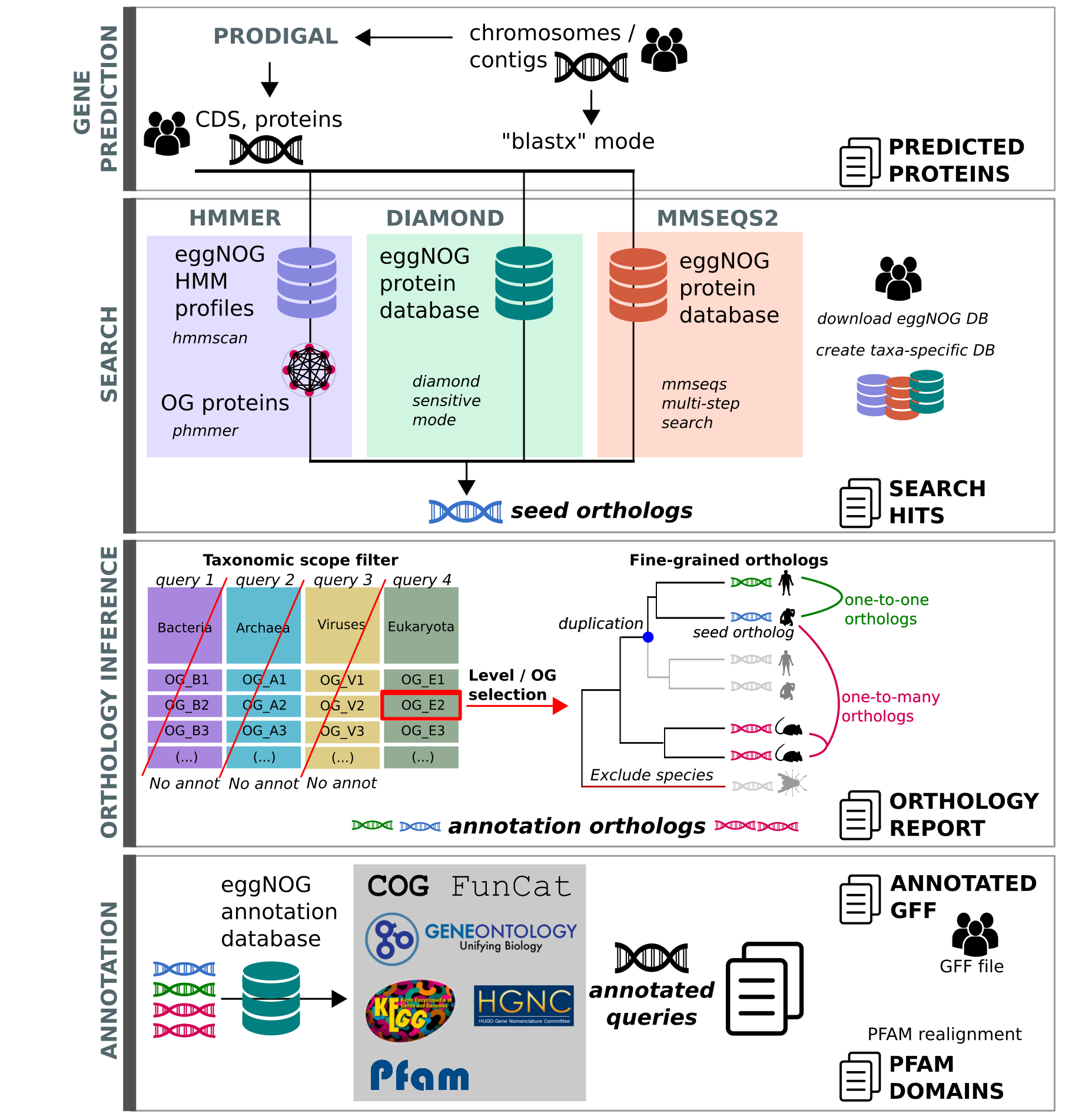
Opis
EggNOG-mapper je alat za brzu funkcionalnu anotaciju novih sekvenci. Koristi unaprijed izračunate ortologne skupine i filogenije iz eggNOG baze podataka (http://eggnog5.embl.de) za prijenos funkcionalnih informacija samo iz fino zrnatih ortologa. Uobičajena upotreba eggNOG-mappera uključuje označavanje novih genoma, transkriptoma ili čak kataloga metagenomskih gena.
Korištenje ortoloških predviđanja za funkcionalnu zabilješku dopušta veću preciznost od tradicionalnih homoloških pretraživanja (tj. BLAST pretraživanja), budući da se izbjegava prijenos zabilješki iz bliskih paraloga (dvostruki geni s većom vjerojatnošću da budu uključeni u funkcionalnu divergenciju).
Verzije
| Verzija | Modul | Supek | Padobran |
|---|---|---|---|
| 2.1.12 | scientific/EggNOG-mapper/2.1.12 |
Primjer korištenja
egg.pbs
#PBS -N eggnog #PBS -l select=1:ncpus=20:mem=10GB #PBS -q cpu cd $PBS_O_WORKDIR module load scientific/EggNOG-mapper/2.1.12 eggnog_mapper.sh emapper.py -i protein.fasta -o test1 --cpu $NCPUS
EggNOG-mapper workflow