Korištenje
Dostupne verzije i pripadajući moduli:
| Verzija | Modul |
|---|---|
| 2.1.1 | bioinfo/parallelfold/2.1.1 |
Parallelfold je modificirana skripta Alphafolda i služi za raščlanjivanje CPU(MSA and template searching) i GPU(prediction model) dijela.
Primjer pokretanja CPU dijela:
cpu_parafold.sge
#!/bin/bash #$ -N parafold2 #$ -pe p28-mpi 10 #$ -cwd module load bioinfo/parallelfold/2.1.1 run_alphafold.sh -d $ALPHAFOLDDB -o output -f -i fasta/actin_gallus1.fasta -p monomer_ptm -t 2021-12-15 --usegpu=False
Bitan je flag –usegpu=False koji naznačuje da se ne koristi GPU neko isključivo CPU i -f koji zaustavlja alphafold nakon kreiranja feautre.pkl u output direktoriju.
Nakon CPU dijela treba pokrenuti GPU dio za dalje. Pokreće se isti job u istom direktoriju, feature.pkl mora biti prisutan u output folderu za GPU dio.
Primjer GPU dijela:
gpu_parafold.sge
#!/bin/bash #$ -N parafold2 #$ -pe gpu 1 #$ -l cores=5 #$ -cwd module load bioinfo/parallelfold/2.1.1 GPUDEVICE=$(cat $TMPDIR/gpu) cuda-wrapper.sh run_alphafold.sh -d $ALPHAFOLDDB -o output -i fasta/actin_gallus1.fasta -p monomer_ptm -t 2021-12-15 -a $GPUDEVICE -n 5
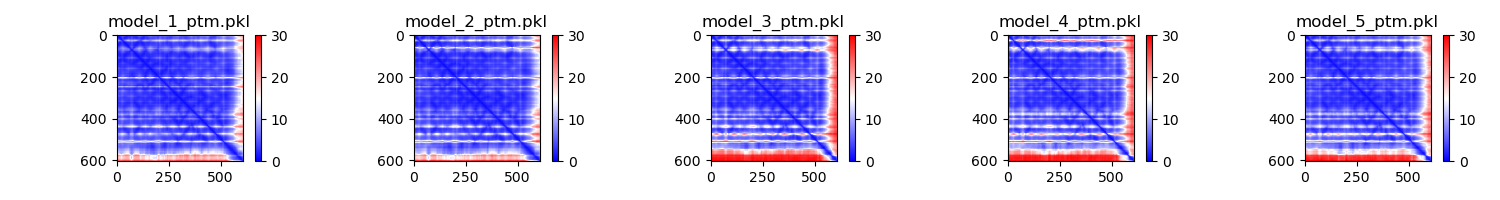
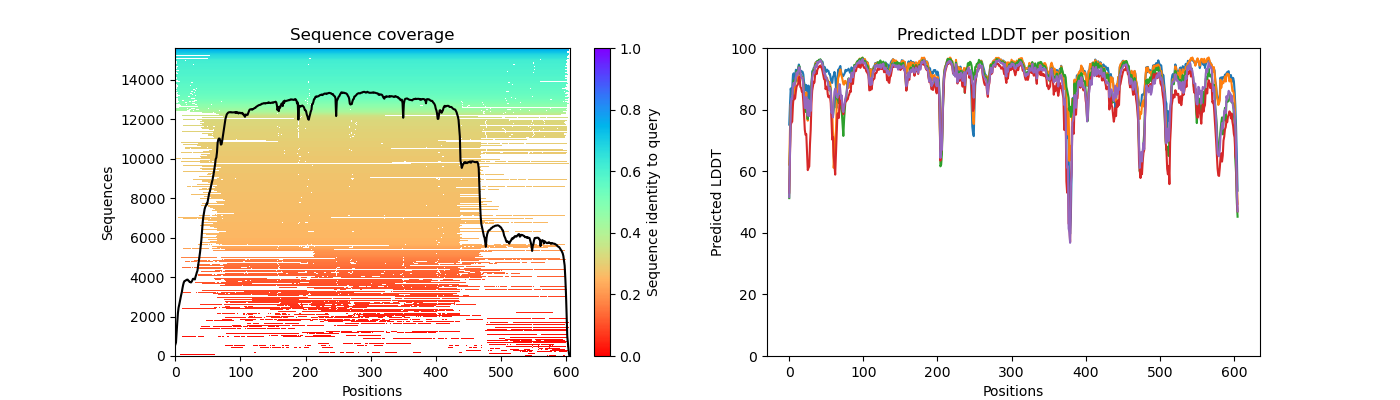
Vizualizacija rezultata PLLDT i PAE
Primjer korištenja:
visualisation.sge
#!/bin/bash #$ -N PLLDT_PAE #$ -pe *mpisingle 2 #$ -cwd module load bioinfo/parallelfold/2.1.1 run_visualisation.sh -i output/Canis_Lupus_dehydrogenase_sub_5/ -o my_outputs -n Canis
Potrebna je putanja do direktorija sa rezultatima za pojedini uzorak (-i), putanja do direktorija gdje će biti stvorene slike(-o), potrebno je i napisati prefix za imena slike(-n)
Rezultati:
Instalacija
Prerequisites
source miniforge3/bin/activate
conda create --prefix /apps/virtenv/alphafold2 python=3.8
conda activate /apps/virtenv/alphafold2
conda install -y -c conda-forge openmm==7.5.1 cudnn==8.2.1.32 cudatoolkit==11.0.3 pdbfixer==1.7
conda install -y -c bioconda hmmer==3.3.2 hhsuite==3.3.0 kalign2==2.04
conda install -y -c conda-forge pandas
pip install absl-py==0.13.0 biopython==1.79 chex==0.0.7 dm-haiku==0.0.4 dm-tree==0.1.6 immutabledict==2.0.0 jax==0.2.14 ml-collections==0.1.0
numpy==1.19.5 scipy==1.7.0 tensorflow==2.5.0
pip install --upgrade jax jaxlib==0.1.69+cuda111 -f https://storage.googleapis.com/jax-releases/jax_releases.html
Install and patch
git clone https://github.com/Zuricho/ParallelFold.gitcd /apps/virtenv/parallelfold2.1.1/lib/python3.8/site-packages/ patch -p0 < ../../../ParallelFold/docker/openmm.patch wget -q https://git.scicore.unibas.ch/schwede/openstructure/-/raw/7102c63615b64735c4941278d92b554ec94415f8/modules/mol/alg/src/stereo_chemical_props.txt